谷歌研究團隊與加州大學聖克魯茲分校的研究人員聯合推出了 DeepSomatic,這是一種創新的人工智能模型,旨在識別癌細胞中的基因變異。在與兒童醫療中心的合作研究中,DeepSomatic 成功發現了其他工具未能識別的10種小兒白血病細胞變異。
DeepSomatic 採用了一種針對癌症基因組的小型變異調用器,能夠兼容 Illumina 短讀段、PacBio HiFi 長讀段和 Oxford Nanopore 長讀段。這一方法在 DeepVariant 的基礎上進行了擴展,能夠檢測單核苷酸變異(SNV)和小插入缺失(indels),支持腫瘤 - 正常和腫瘤單一工作流程,包括福爾馬林固定石蠟包埋(FFPE)模型。
DeepSomatic 的工作原理是將對齊後的讀段轉換爲圖像般的張量,這些張量編碼了堆疊情況、基礎質量和對齊上下文。通過卷積神經網絡,模型對候選位點進行分類,判定其爲體細胞變異或非變異,最終生成 VCF 或 gVCF 文件。這種設計使得 DeepSomatic 在技術平臺上具有高度的適應性,因爲該張量能夠總結不同技術間的局部單倍型和錯誤模式。
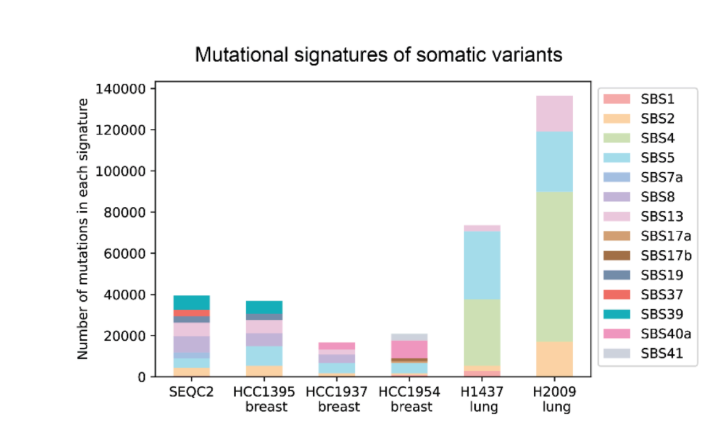
在數據集和基準測試方面,DeepSomatic 使用 CASTLE(癌症標準長讀段評估)數據集進行訓練和評估。該數據集包含6對匹配的腫瘤和正常細胞系,使用 Illumina、PacBio HiFi 和 Oxford Nanopore 進行了全基因組測序。研究團隊發佈了基準集和訪問權限,以便其他研究者進行重用,這填補了多技術體細胞訓練和測試資源的空白。
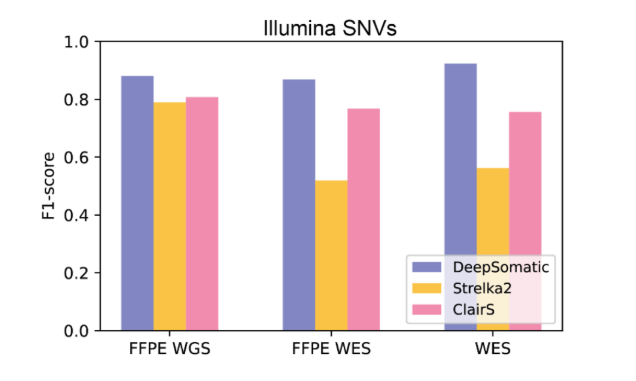
結果顯示,DeepSomatic 在單核苷酸變異和小插入缺失的檢測上都優於目前廣泛使用的方法。例如,在 Illumina 測序的插入缺失方面,DeepSomatic 達到了約90% 的 F1分數,而其他方法僅爲80%;在 PacBio 測序中,DeepSomatic 的 F1分數超過80%。研究團隊還報告了329,011個體細胞變異的發現,進一步驗證了 DeepSomatic 在插入缺失檢測方面的強大能力。
研究:https://research.google/blog/using-ai-to-identify-genetic-variants-in-tumors-with-deepsomatic/
劃重點:
🌟 DeepSomatic 能夠識別多種癌細胞基因變異,涵蓋多種測序平臺。
🔍 該模型採用卷積神經網絡,將讀取信息轉化爲圖像張量,確保準確性與一致性。
📊 在基準測試中,DeepSomatic 的檢測精度明顯優於現有主流方法,尤其是在插入缺失的檢測上。
