グーグルの研究チームとカリフォルニア大学サクラメント校の研究者たちは、がん細胞における遺伝子変異を特定するための革新的な人工知能モデル「DeepSomatic」を共同で開発しました。児童医療センターとの共同研究では、DeepSomaticは他のツールでは検出できなかった10種類の小児白血病細胞の変異を成功裏に発見しました。
DeepSomaticは、がんゲノム向けに設計された小さな変異呼び出し器を使用しており、Illuminaの短読長、PacBio HiFiの長読長、Oxford Nanoporeの長読長に対応しています。この方法はDeepVariantを基盤としており、単一ヌクレオチド多型(SNV)および小さな挿入・欠損(indels)の検出が可能で、腫瘍-正常対応および腫瘍単一ワークフローをサポートし、フォルマリン固定パラフィン包埋(FFPE)モデルにも対応しています。
DeepSomaticの仕組みは、アライメント後のリードを画像のようなテンソルに変換し、それらのテンソルはスタック状態、ベース品質、およびアライメントの文脈を符号化します。畳み込みニューラルネットワークにより、候補位置を体細胞変異または非変異として分類し、最終的にVCFまたはgVCFファイルを生成します。この設計により、異なる技術間のローカルハプロタイプやエラーのパターンを要約できるため、DeepSomaticは技術プラットフォームに対して高い適応性を持っています。
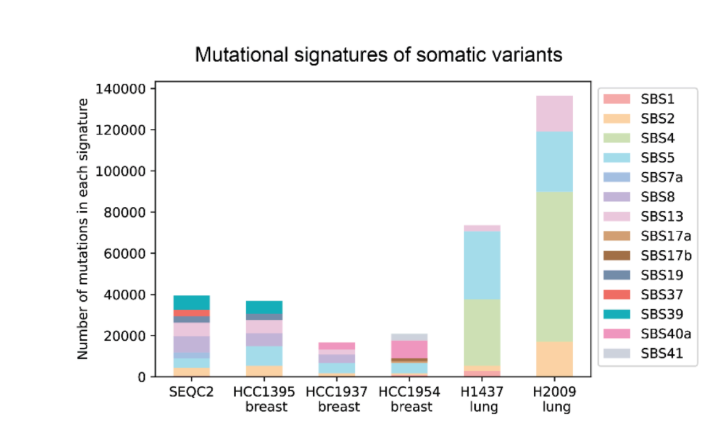
データセットとベンチマークテストに関しては、DeepSomaticはCASTLE(がん標準長読長評価)データセットを訓練および評価に使用しています。このデータセットには、Illumina、PacBio HiFi、Oxford Nanoporeを使用して全ゲノムシーケンスされた6つのマッチングされた腫瘍および正常細胞系が含まれています。研究チームはベンチマークセットとアクセス権を公開し、他の研究者が再利用できるようにしました。これにより、複数技術での体細胞トレーニングとテストリソースの空白が埋められました。
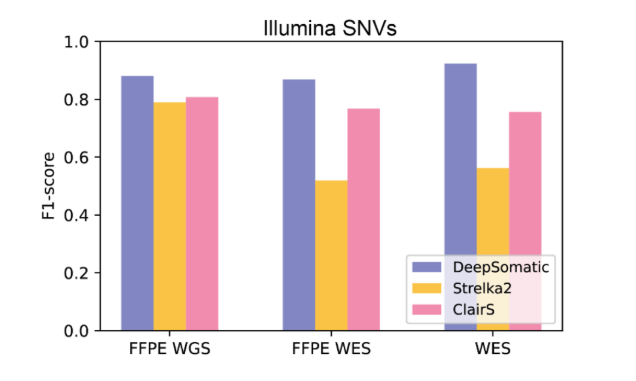
結果によると、DeepSomaticは現在広く使用されている手法よりも単一ヌクレオチド多型および小さな挿入・欠損の検出において優れています。例えば、Illuminaシーケンスの挿入・欠損では、DeepSomaticは約90%のF1スコアを達成し、他の手法はわずか80%にとどまりました。PacBioシーケンスでは、DeepSomaticのF1スコアは80%を超えました。また、研究チームは329,011個の体細胞変異の発見を報告し、DeepSomaticが挿入・欠損検出において強力な能力を持っていることをさらに確認しました。
研究:https://research.google/blog/using-ai-to-identify-genetic-variants-in-tumors-with-deepsomatic/
ポイント:
🌟 DeepSomaticは、さまざまながん細胞の遺伝子変異を識別し、多数のシーケンスプラットフォームに対応しています。
🔍 このモデルは畳み込みニューラルネットワークを使用し、リード情報を画像のようなテンソルに変換することで、正確性と一貫性を確保しています。
📊 ベンチマークテストにおいて、DeepSomaticは既存の主流手法よりも検出精度が高く、特に挿入・欠損の検出において顕著です。
